随着信息科学的迅猛发展,计算机辅助的药物-靶标相互作用预测方法作为一种新兴的药物靶标识别手段,在新药研发源头创新中发挥了重要的作用。针对药物-靶标相互作用预测体系的建立,引入基因组学、蛋白组学和药理学等异构数据可以提高稀疏数据集上的预测精度,但如何系统且高效地整合大规模异构数据仍是当前的研究难点。
近日,浙江大学智能创新药物研究院人工智能制药平台侯廷军主任团队联合浙江大学控制学院贺诗波团队、中南大学曹东升团队和腾讯量子实验室,针对药物-靶标相互作用预测方法开发中大规模异构数据整合难点,提出了一种基于知识图谱和推荐系统的药物靶标相互作用预测新方法KGE_NFM。该方法高效地整合多组学的信息,为全新药物-靶标相互作用对的发现提供了功能强大的计算工具。相关工作以标题为“A unified drug-target interaction prediction framework based on knowledge graph and recommendation system”发表于国际权威期刊《自然·通讯》(Nature Communications)。

为了解决传统方法中存在的数据稀疏性、冷启动问题,平台团队通过知识图谱对可利用的异构数据进行建模,无需节点间的相似性计算,简单高效地整合了不同来源的组学数据。在基于知识图谱的信息表征基础上,该方法结合推荐系统方法--神经因子分解机进行药物靶标相互作用预测,有效去除了复杂生物网络中的噪声,并整合药物和蛋白结构信息的传统表征方式,进一步保证在真实场景下高精度,高稳定性的预测性能。
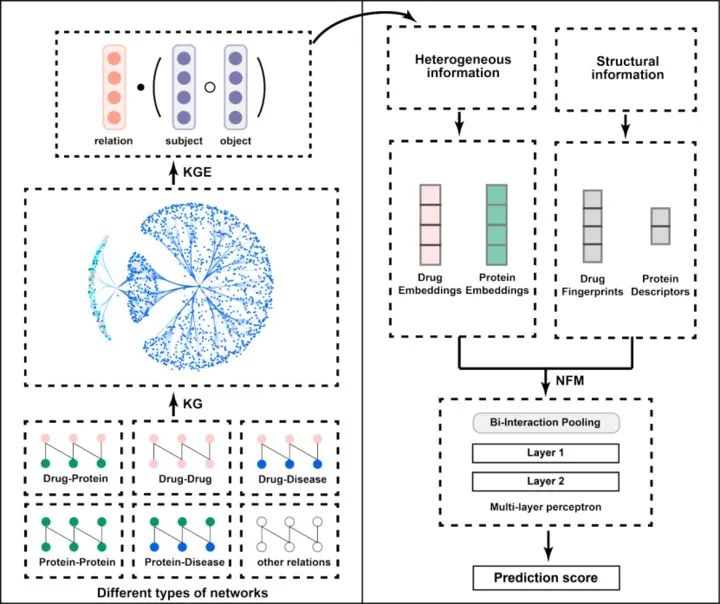
图 KGE_NFM方法的工作流程图
在三类贴近真实应用的测试场景中,KGE_NFM均保持了高精度的预测性能,尤其是在对于靶标蛋白的冷启动场景下,KGE_NFM在四个基准数据集上相比于传统方法有着15%~30%的预测精度的提升。结果表明KGE_NFM是一种极具竞争力的药物-靶标相互作用预测方法,有望促进复杂疾病蛋白质靶点的发现和分子作用机制的阐明。
浙江大学智能创新药物研究院着力打造人工智能驱动的创新药物发现、设计与合成、筛选与评价的一体化新药创制研发体系,在基于靶标结构的虚拟筛选、基于分子模拟的靶标-配体识别机制研究、药物ADMET和类药性质的理论预测、基于药物靶点结构的药物设计和筛选、基于AI的多组学数据表征和药效预测等方面开发了多个算法和数据库。基于药物靶标相互作用预测开发的算法也将进一步加速人工智能赋能的新药研发,助推创新药物发现。


